Hoe AI nu ook ‘chaos’ in eiwitten tot orde brengt
Wetenschappers hebben een nieuwe machine-learningmethode ontwikkeld die het mogelijk maakt om zogeheten intrinsiek gedesordeerde eiwitten (IDP’s) te ontwerpen — eiwitten zonder vaste, stabiele vorm. Die aanpak kan helpen om beter te begrijpen welke rol die flexibele eiwitten spelen in cellen, en misschien leiden tot nieuwe therapeutische toepassingen.
De uitdaging van gedesordeerde eiwitten
In de conventionele wereld van eiwitten geldt: structuur bepaalt functie. Eiwitten vouwen zich netjes in driedimensionale vormen, en voorspellen van die vorm is de sleutel om hun functie te begrijpen. Denk aan AI-systemen zoals AlphaFold, die zeer succesvol zijn in structurele voorspellingen.
|
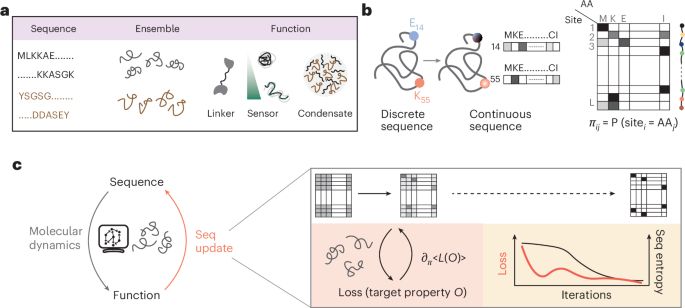
Generalized design of sequence–ensemble–function relationships for intrinsically disordered proteins - Nature Computational ScienceThe design of folded proteins has advanced substantially in recent years. However, many proteins and protein regions are intrinsically disordered and lack a stable fold, that is, the sequence of an intrinsically disordered protein (IDP) encodes a vast ensemble of spatial conformations that specify its biological function. |
Maar zo’n aanpak faalt wanneer het eiwit géén stabiele vorm aanneemt — bij intrinsiek gedesordeerde eiwitten. Ongeveer 30 % van het menselijke proteoom bestaat uit IDP’s, die zich constant bewegen en geen vaste vorm innemen.
Die dynamiek maakte dat IDP’s lange tijd buiten bereik lagen van AI-ontwerpen: er was geen vaste structuur om op te mikken.
De nieuwe benadering: orde in de chaos
Onderzoekers aan Harvard’s SEAS en Northwestern ontwikkelden een methode die automatisch differentiëren (een wiskundige truc uit de wereld van deep learning) combineert met moleculaire dynamica-simulaties.
In plaats van te vertrouwen op enorme datasets om patronen te leren, gebruikt de aanpak fysica en optimalisatie: eiwitsequenties worden stap voor stap aangepast en getest in simulaties, waarbij het algoritme leert welke kleine wijzigingen leiden tot gewenste eigenschappen.
Met andere woorden: het systeem functioneert als een “zoekmachine” voor aminozuurcombinaties die het gedrag van een flexibel eiwit sturen.
|
Beyond Folding: Machine Learning Designs Intrinsically Disordered ProteinsPhysics-based ML framework designs IDPs—biomolecules without fixed structures that underlie key functions and diseases such as Parkinson’s. |
Wat bereikt dit?
Met deze methode kunnen onderzoekers IDP’s ontwerpen met specifieke eigenschappen — bijvoorbeeld flexibiliteit, bindingsdynamiek of respons op omgevingsveranderingen. Zo kunnen ze beter onderzoeken wat IDP’s doen in de cel, en hoe ze bijdragen aan ziektes als Parkinson.
Bovendien opent het de deur voor nieuw eiwit-engineering buiten de klassieke, “stevige” eiwitten om — een stap die van belang kan zijn in biotechnologie en medicijnontwikkeling.
Blik op de toekomst
Hoewel dit een spectaculaire stap is, liggen er ook uitdagingen: het simuleren van gedesordeerde toestanden vereist grote rekencapaciteit en nauwkeurige fysische modellen. Verder is het een kunst om het juiste evenwicht te vinden tussen automatisering en biologische complexiteit.
Toch groeit het veld snel. Nieuwe algoritmen zoals ALBATROSS, IDPForge en andere benaderingen proberen de sequentie-naar-ensemble-eigenschappen van IDP’s op schaal te voorspellen.
Als deze tools volwassen worden, zou het mogelijk zijn om op maat gemaakte flexibele eiwitten te “programmeren” voor toepassingen van diagnostiek tot therapie — eiwitten die juist functioneren dankzij hun flexibiliteit, niet ondanks die.
Met hun nieuwe methode tonen de onderzoekers dat zelfs in de ogenschijnlijke wanorde van gedesordeerde eiwitten, patronen te ontdekken én te sturen zijn. De stap van ‘vouwen’ naar ‘ontwerpen’ van eiwitten zonder vaste structuur markeert een boeiende doorbraak in de interface tussen biologie, fysica en AI.